![]()
Aim of our research / Backgroung of our research
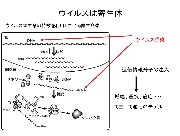 Virus is non-living thing but it can multiply and propagate its progeny via specific biological processes called the infection cycle. The production processes of virus depend on the transcription and translation mechanisms of living host cells. We can find so many viruses that use RNA molecule as their genetic information. Since RNA molecules are not stable chemically, it seems to be reasonable that all living cells utilize DNA molecule as their main genetic information. Moreover, RNA molecules usually exist as a single-strand form and their replication is sure to be error-prone because there is no proofreading system during RNA replication. Therefore, infection cycle of RNA virus will inevitably produce so many mutants of its original. Why RNA virus can survive against such defects? One might consider that the error-prone replication of RNA possibly supports the speed of an adaptive evolution of virus. The other might think that error does not function and the adsorption process of infection cycle works as an efficient quality control check and only infectious virus succeeds to propagate. We want to understand the meaning of every step of RNA virus infection cycle under a physiological condition. For this purpose, bacteria and bacteriophage system appears to be convenient. As a host bacteria, Escherichia coli is easy to propagate in a laboratory and its genome and phenotype are both well-known and suitable mutant strains will be available when we wish to use. On the other hand, as an RNA bacteriophage, we use Qbeta phage. Qbeta phage is one of the most famous RNA bacteriophage which contains a single-strand RNA genome (4217 nt) in its protein shell. There are 4 kinds of proteins encoded on the phage genome, e.g. coat protein, a main subunit of RNA replicase, and two maturation proteins. Phage research sounds old and bored because so many historical works have been done since early 20th century. But most of our knowledge are not quantitatively presented. We want to understand precisely the basic strategy of RNA phage to survive and we also hope to innovate the ways to utilize the host specificity, adaptability and evolvability of phage.
Virus is non-living thing but it can multiply and propagate its progeny via specific biological processes called the infection cycle. The production processes of virus depend on the transcription and translation mechanisms of living host cells. We can find so many viruses that use RNA molecule as their genetic information. Since RNA molecules are not stable chemically, it seems to be reasonable that all living cells utilize DNA molecule as their main genetic information. Moreover, RNA molecules usually exist as a single-strand form and their replication is sure to be error-prone because there is no proofreading system during RNA replication. Therefore, infection cycle of RNA virus will inevitably produce so many mutants of its original. Why RNA virus can survive against such defects? One might consider that the error-prone replication of RNA possibly supports the speed of an adaptive evolution of virus. The other might think that error does not function and the adsorption process of infection cycle works as an efficient quality control check and only infectious virus succeeds to propagate. We want to understand the meaning of every step of RNA virus infection cycle under a physiological condition. For this purpose, bacteria and bacteriophage system appears to be convenient. As a host bacteria, Escherichia coli is easy to propagate in a laboratory and its genome and phenotype are both well-known and suitable mutant strains will be available when we wish to use. On the other hand, as an RNA bacteriophage, we use Qbeta phage. Qbeta phage is one of the most famous RNA bacteriophage which contains a single-strand RNA genome (4217 nt) in its protein shell. There are 4 kinds of proteins encoded on the phage genome, e.g. coat protein, a main subunit of RNA replicase, and two maturation proteins. Phage research sounds old and bored because so many historical works have been done since early 20th century. But most of our knowledge are not quantitatively presented. We want to understand precisely the basic strategy of RNA phage to survive and we also hope to innovate the ways to utilize the host specificity, adaptability and evolvability of phage.
Main stream of our research
1.Infection dynamics of bacteriophage
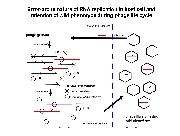
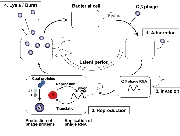
 Qbeta phage is one of the most famous bacteriophage in nature. "Bacteriophage" is certain kind of virus that adsorb to the bacterial cell surface (or pilus), inject its genome into bacterial cell and propagate the phage progeny by embezzling both the translation system that work for the production of the host proteins and the metabolic products (ribonucleoside and amino acids) used for the RNA replication and protein synthesis. Qbeta phage usually propagates in Gram-negative bacterial cell e.g. Escherichia coli (E. coli). Genome molecule of Qbeta phage is one, single-strand RNA (4217 nt). Once the genome RNA invades into the host cell, the genome functions as an mRNA for the phage-derived proteins, e.g. RNA-dependent RNA replicase (Qbeta replicase) and coat protein and so on. Reaction of Qbeta replicase has high specificity and this RNA polymerase strictly selects phage RNA as a template molecule for replication.
Qbeta phage is one of the most famous bacteriophage in nature. "Bacteriophage" is certain kind of virus that adsorb to the bacterial cell surface (or pilus), inject its genome into bacterial cell and propagate the phage progeny by embezzling both the translation system that work for the production of the host proteins and the metabolic products (ribonucleoside and amino acids) used for the RNA replication and protein synthesis. Qbeta phage usually propagates in Gram-negative bacterial cell e.g. Escherichia coli (E. coli). Genome molecule of Qbeta phage is one, single-strand RNA (4217 nt). Once the genome RNA invades into the host cell, the genome functions as an mRNA for the phage-derived proteins, e.g. RNA-dependent RNA replicase (Qbeta replicase) and coat protein and so on. Reaction of Qbeta replicase has high specificity and this RNA polymerase strictly selects phage RNA as a template molecule for replication.
Generally, there is no proof-readind mechanism during RNA-dependent RNA polymeration by Qbeta
replicase, replicated RNA molecules should have many errors in their sequence and further replication might cause the original RNA.
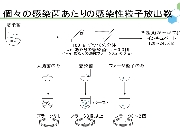
一般にRNA複製反応には校正機構が無く、高い変異導入率を有するため、ファージゲノムの複製段階では高いerror rateにより自身の増幅が邪魔される筈である。実際、大腸菌を宿主とするRNAウイルスであるQβファージのゲノムは4200塩基ほどの一本鎖RNAだが、報告されている突然変異率は6.5(/genome/generation)と報告されている(Drake (1993) PNAS 90、4171-4175.)。もし鋳型となるRNAゲノムが正しい塩基配列であったとしても、このファージゲノムからたった1回のコピー(複製反応)でつくられたRNA中にはすでに平均6.5個の塩基置換が起きていることを意味する。しかし、ファージは野生型形質を失うことなく植え継がれる。ファージの増幅には、大腸菌への吸着(F繊毛上の受容体認識)、 RNAゲノムの注入、ゲノムRNAをmRNAとした大腸菌内での複製酵素翻訳、RNAゲノムの増幅、ファージ粒子構成タンパク質による複製酵素翻訳過程の阻害、溶菌タンパク質(A2)の発現、大腸菌内でファージ粒子の再構築、大腸菌の溶菌を伴う放出、という多段階かつ一連の流れがあり、どの過程が失敗してもファージ粒子の増幅は達成されない。この複雑な調節系を大腸菌内という変化し続ける(そして相互作用・生体防御を発動する)環境で自身の増幅を制御できたQβファージのRNAゲノムのみが、ファージ粒子に包まれ植え継がれるため、上記の「遺伝子発現調節」が淘汰圧となって「野生型」表現型を有する遺伝子型群が生き残るであろう。
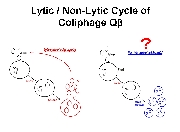
一方、F+もしくはHfr形質の大腸菌の対数増殖期にQβファージを感染させた後にこれを希釈し培養すると、大腸菌とファージ粒子の双方が共存して増殖する様子が確認され、この共培養は抗Qβファージ血清存在下でも成立することが1980年頃渡辺格らによって報告されている (Watanabe I et al. (1979) Microbiol. Immunol. 23, 1077-1083)。これは、感染菌を溶かして子孫を外界に放出させるQβファージの感染サイクルとは全く異なる増殖戦略であり、Qβファージの「擬似溶原化状態」を報告したものである。in situ hybridizationの結果、タンパク質翻訳までの過程が進行した状態でとどまった感染状態であることが示唆されており、何らかの理由でファージタンパク質の発現・翻訳・蓄積が停止した状態で保持されたものではないか、と考えられている。野生型ファージ用の植え継ぎにより得られるファージゲノム(溶菌活性など毒性の高い形質)と大腸菌内に留まりやすいファージゲノム(毒性の低い形質)を得た場合、そこには如何なる適応変異の差が読み取られるであろうか?
2.新規ウィルス研究法の開発/ Improvement of procedures for virus research
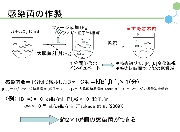
 ウイルスやファージを理解するだけでなく、その利用法をも考える場合、具体的かつ定量的に増殖戦略を解明することが必要である。われわれは定量的に説得力の高い研究手法を適宜提案し、ファージと大腸菌の感染ダイナミクスに関する研究を進める。そのために、大腸菌の数を測定する方法として便利なコロニー計数法や、ファージの数を測定する方法としてよく用いられてきたプラークアッセイ法も検討対象とし、これらが不適当な場合には他の計数法を用いている。また、1950年代からのファージ研究の歴史の中で提案されてきた他の手法に関しても、定量性の観点から再検証し、適切な工夫を随時盛り込むことによって新たな研究手法として提案していく。
ウイルスやファージを理解するだけでなく、その利用法をも考える場合、具体的かつ定量的に増殖戦略を解明することが必要である。われわれは定量的に説得力の高い研究手法を適宜提案し、ファージと大腸菌の感染ダイナミクスに関する研究を進める。そのために、大腸菌の数を測定する方法として便利なコロニー計数法や、ファージの数を測定する方法としてよく用いられてきたプラークアッセイ法も検討対象とし、これらが不適当な場合には他の計数法を用いている。また、1950年代からのファージ研究の歴史の中で提案されてきた他の手法に関しても、定量性の観点から再検証し、適切な工夫を随時盛り込むことによって新たな研究手法として提案していく。
3.モデル生命システムの共進化/ Experimental model analysis for understanding co-evolution mechanisms of life
生物は多かれ少なかれ他の存在物(生物・無生物問わず)と相互作用しながら、生存してきた。ある報告によると、地球上には1000000000000000000000000000000(0が30個)のバクテリアとそれとほぼ同数のファージが生息している惑星であるらしい。実験室において、バクテリアとファージの共存培養を行い、そのダイナミクスを両者の軍拡競争として追跡することで、生物間の相互作用を定量的に観察できる。特にバクテリアとファージの共存培養軍拡競争的な共進化のダイナミクスが観察できるであろう。バクテリアがひとたび過度にファージ耐性を獲得すると、ファージは感染や増殖ができなくなってしまう。一方ファージも適度に感染力を低く保たないと、大腸菌を全滅させてしまい、結果としてファージは感染や増殖ができなくなってしまう。バクテリアとファージの共存培養軍拡競争的な共進化はどのようなシナリオで進行する性質があるのだろうか。

 Home
Home Research
Research Publications
Publications Gallery
Gallery